
论文基本信息
- 标题: Universal and extensible language-vision models for organ segmentation and tumor detection from abdominal computed tomography
- 中文标题: 通用且可扩展的语言-视觉模型,用于腹部CT中的器官分割和肿瘤检测
- 发表年份: 2024年6月
- 期刊/会议: Medical Image Analysis
- 分区/IF: SCI 1区 / 10.7
- 作者: Jie Liu; Yucheng Tang, Zongwei Zhou (一作;通讯)
- 单位: 香港城市大学
- DOI: https://doi.org/10.1016/j.media.2024.103226
- 开源代码: https://github.com/ljwztc/CLIP-Driven-Universal-Model
摘要
人工智能(AI)在器官分割和肿瘤检测方面的进展。然而,这些AI模型常常在处理部分标注数据集的灵活性和面对新类别的可扩展性方面存在困难,主要是由于独热编码(one-hot)、架构设计和学习方案的限制。为了克服这些限制,我们提出了一个通用、可扩展的框架,使得单一模型—被称为通用模型(Universal Model)——能够处理多个公共数据集,并适应新的类别(例如器官/肿瘤)。
首先,我们引入了一种新颖的语言驱动参数生成器,利用来自大型语言模型的语言嵌入,相比于独热编码,丰富了语义编码。
其次,我们用轻量级的、类别特定的头部替代传统的输出层,使得通用模型能够同时分割25种器官和6种类型的肿瘤,并便于添加新的类别。
我们在由14个公共数据集组成的3410个CT上训练我们的通用模型,然后在来自四个外部数据集的6173个CT上进行测试。通用模型在医学分割十项全能赛公共排行榜上取得了六个CT任务的第一名,并在 BTCV 数据集上表现卓越。
总之,通用模型表现出了显著的计算效率(比其他特定数据集模型快6倍),展示了在不同医院之间的强大泛化能力,良好地迁移到多个下游任务上,更重要的是,在减少先前学习类别遗忘的同时,促进了对新类别的可扩展性。代码、模型和数据集可以在 https://github.com/ljwztc/CLIP-Driven-Universal-Model 获取。
Introduction
尽管当前数据集中普遍存在肝脏的标注,但肝脏叶的划分仍未受到充分研究,这可能导致未来肝脏叶与整体肝脏之间的标注重叠。为满足这些新兴需求,最新的工作包括利用人在环中重新标注现有数据集,并相应地重新训练模型。然而,这种方法会带来显著的标注成本,特别是对于三维医学成像,以及从头重新训练模型的大量计算消耗。因此,探索一种新的框架至关重要,能有效处理新的器官/肿瘤注释,同时减少与重新训练模型相关的计算负担。
设计这样一个持续多器官分割和肿瘤检测框架时,需要解决几个挑战。
第一个挑战涉及编码正交性问题。现有方法通常采用独热编码,忽略器官和肿瘤之间的语义关系。例如,当使用独热编码表示肝脏 [1,0,0]、胰腺 [0,1,0] 和胰腺肿瘤 [0,0,1] 时,这些类别之间缺乏语义上的区别。一种可能的解决方案是少热编码(few-hot),其中肝脏、胰腺和胰腺肿瘤可以分别编码为 [1,0,0]、[0,1,0] 和 [0,1,1]。尽管少热标签可以表明胰腺肿瘤是胰腺的一部分,但器官之间的关系仍然是正交的。此外,当向当前框架添加新的器官时,这些编码方法变得难以扩展,因为整个编码方案需要修改以适应新的类别。
第二个挑战在于标签分类的不一致性,公开可用的数据集通常包含不同类别的注释。这种标签空间的差异限制了传统的分割头部,该头部采用预定义的核数和Softmax激活函数,基于固定的类别为每个体素生成单一类别的预测(Liu等人,2022a;Ma等人,2021;Schoppe等人,2020)。因此,它缺乏灵活性来适应新的器官/肿瘤注释和处理详细的解剖标注。例如,“右叶肝”是“肝脏”的一部分,“肾脏肿瘤”代表“肾脏”的一个子体积,这意味着某些体素不完全属于单一特定的类别,符合临床需求。一个简单的解决方案是替换分割头部并从头开始重新训练,但这会遗忘已学习的知识并导致计算消耗。这促使我们设计一个新颖的分割头部框架,允许灵活预测并对新类别进行扩展。
第三个挑战涉及设计用于持续学习的动态网络架构。在注释新的器官/肿瘤时,医学分割模型应能够在不访问先前数据的情况下扩展其分割能力,以遵守医疗隐私法规。现有工作通常冻结编码器和解码器,并在学习新类别时添加额外的解码器(Ji等人,2023),导致网络参数的巨大内存成本。此外,新添加的解码器缺乏对新添加的器官/肿瘤与现有类别之间关系的认识,从而限制了对现有知识的利用。因此,我们的目标是设计一个网络架构,在不同的持续学习步骤中最小化参数的添加,并确保对新添加的器官/肿瘤与现有类别之间关系的认知。
方法简述
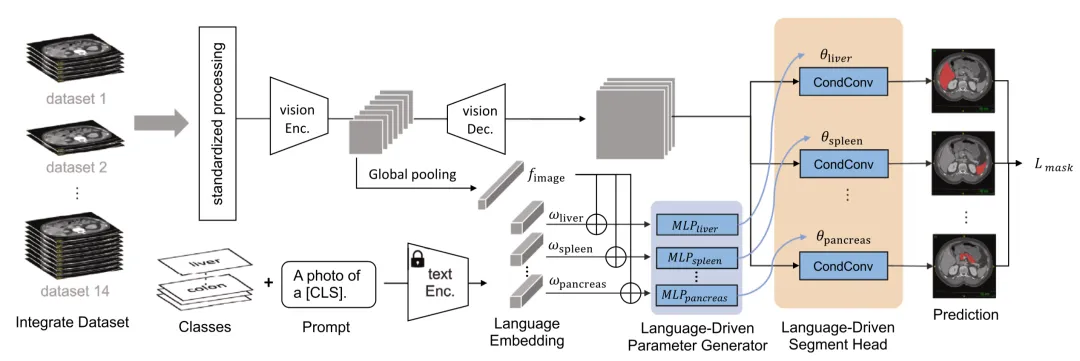
我们的网络架构由两个主要部分组成:语言分支和视觉分支。语言分支利用大型语言模型(如CLIP)生成每个器官或肿瘤的语言嵌入,帮助模型理解器官和肿瘤之间的语义关系。视觉分支采用Swin UNETR架构,这是一种基于Transformer的模型,能够有效提取CT图像中的特征信息。在工作流程中,语言分支首先生成每个器官或肿瘤的语言嵌入,同时视觉分支提取CT图像的特征。然后,这些语言嵌入和图像特征结合起来,生成一个统一的表示。最后,通过特定的分割头,根据结合后的特征生成每个器官或肿瘤的二值掩码,从而实现对图像中每个像素的分类。在持续学习过程中,当有新的器官或肿瘤出现时,我们会添加新的模块来处理这些新类,而不会影响到模型对旧类的处理。同时,我们使用伪标签技术保留旧类的知识,防止模型遗忘之前学到的内容。通过这种结构,模型能够灵活应对多种数据集和新的分割任务,同时保持高效和准确。
实验设计
数据集
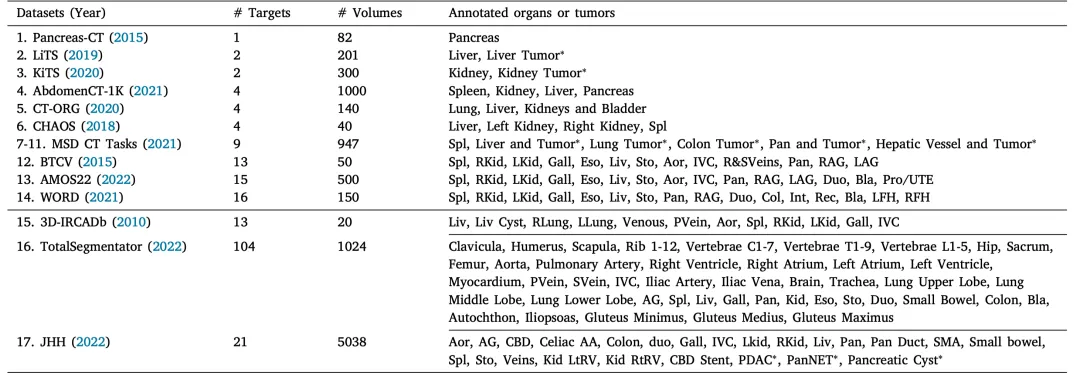
表 1 (下图)列出了用于开发通用模型的14个公开数据集和3个独立验证数据集的详细信息。这些数据集包含不同的器官和肿瘤标注,并且体积大小各不相同
性能比较
为了评估我们方法的有效性,我们在医学分割十项全能(MSD)数据集的官方测试集和五折交叉验证中进行了全面评估。表3详细比较了我们模型在各种指标上的性能,
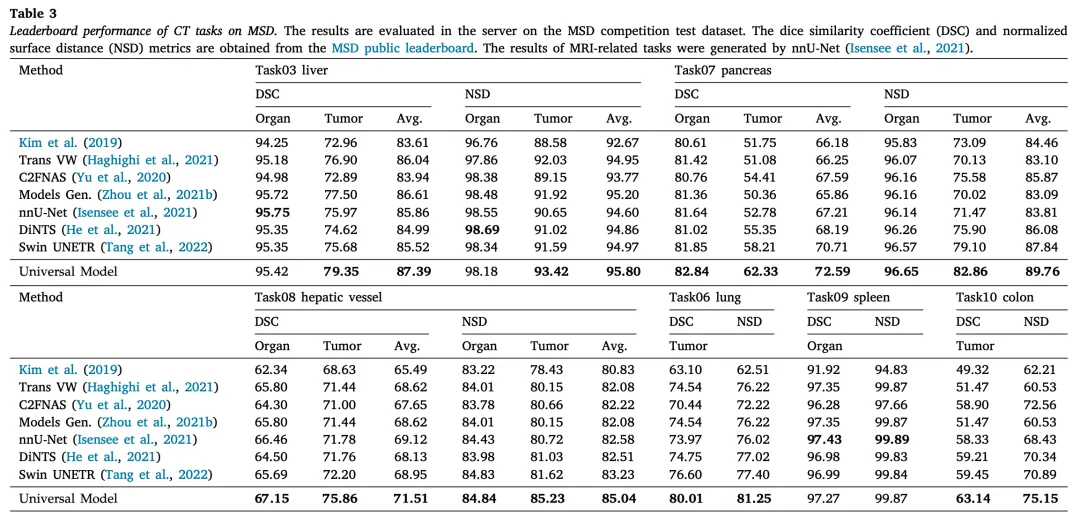
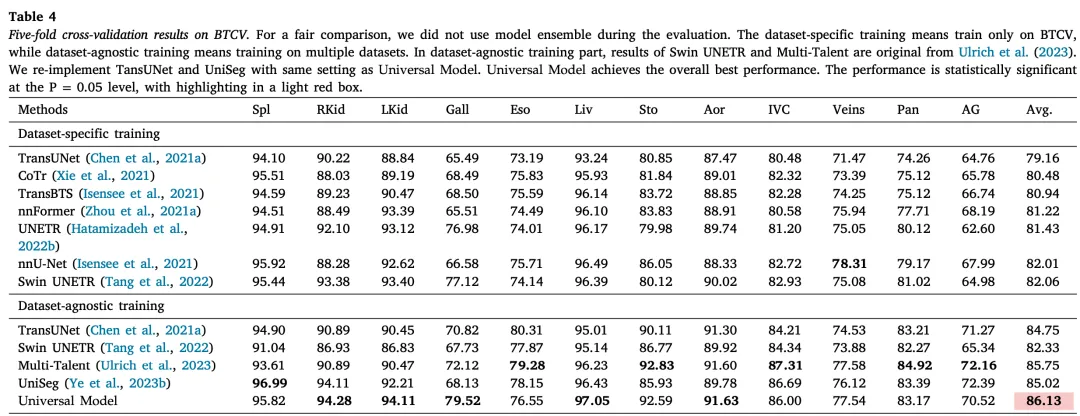
表4展示了通用模型与其他方法在BTCV基准下的数据集特定训练和数据集不可知训练中的比较:
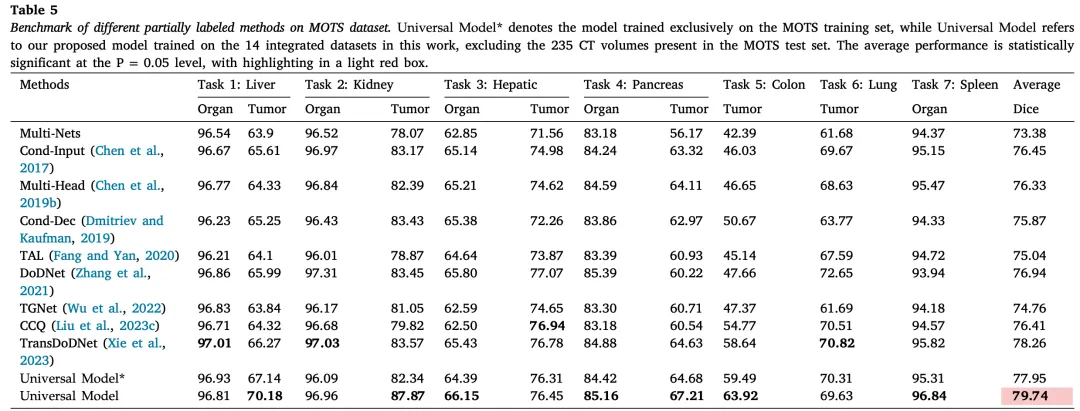
为了公平比较通用模型与其他部分标签学习方法,我们使用多器官和肿瘤分割(MOTS)数据集(Zhang等人,2021)作为基准。表5展示了我们提出的框架的有效性,与其他最先进的方法相比,通用模型取得了第二好的性能
新类别扩展结果
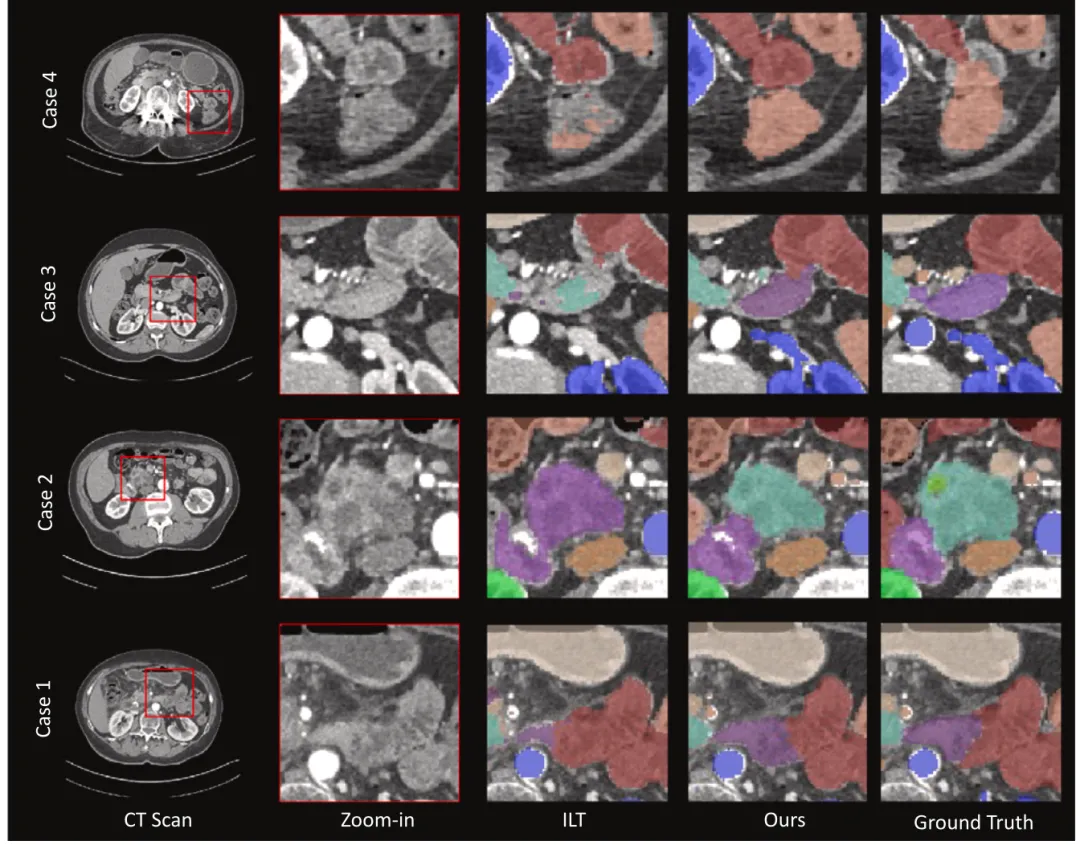
为了提供定性的评估,我们在图6中展示了我们提出的方法与最佳基线方法ILT在身体扩展基准测试中的分割结果。例如,在案例2中,ILT在区分胃和胰腺时出现了混淆。相反,我们的模型由于采用了独立预测的LPG和CSH技术,能够准确区分这两个器官,避免了不同器官之间的矛盾。该可视化有效地展示了我们的方法在器官分割中始终取得了正确的结果。
推理速度
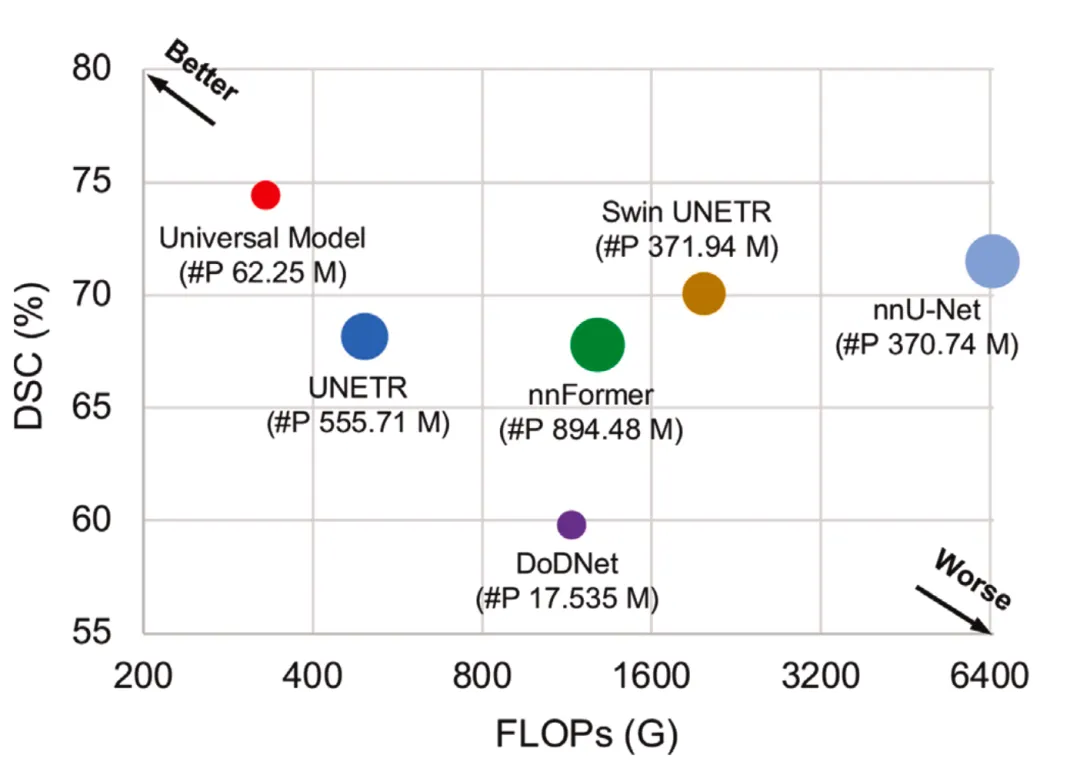
该图展示了在六个医学分割十项全能(MSD)任务中,平均DSC相似系数(DSC)得分与每秒浮点运算次数(FLOPs)之间的关系
总结
这篇文章提出了一种新颖的通用语言-视觉模型,用于腹部CT图像的器官分割和肿瘤检测。通过整合语言嵌入和分割模型,研究人员开发了一个灵活且强大的框架,能够处理多种数据集和新的类别,同时最大限度地减少对先前学习知识的遗忘。
文章代码已开源。